就有人问,生信的文章能发到多少分?如果你是像华科薛宇教授一样的大牛,弄一套算法,编一个生信分析工具,十几分妥妥的,引用量杠杠的。但是,那是大牛,一般来说,按「常规套路」出牌的这种生信分析文章分值在 0-2 分之间。但也有些不做实验的生信分析文章能发到个 4-5 分,那么生信分析的文章怎么样能达到一个比较高的层次呢?
这里,我们给大家分享两篇文章来说一说一些进阶的文章思路,一篇是发表在我们的老朋友「Oncotarget」上的,另一篇是发表在「Journal of Proteome Research」(IF = 4.1)上的。
先看 Oncotarget 这篇「Genomic expression differences between cutaneous cells from red hair color individuals and black hair color individuals based on bioinformatic analysis」,文章是做的黑色素瘤的两种不同表型的个体的差异基因的生信分析。
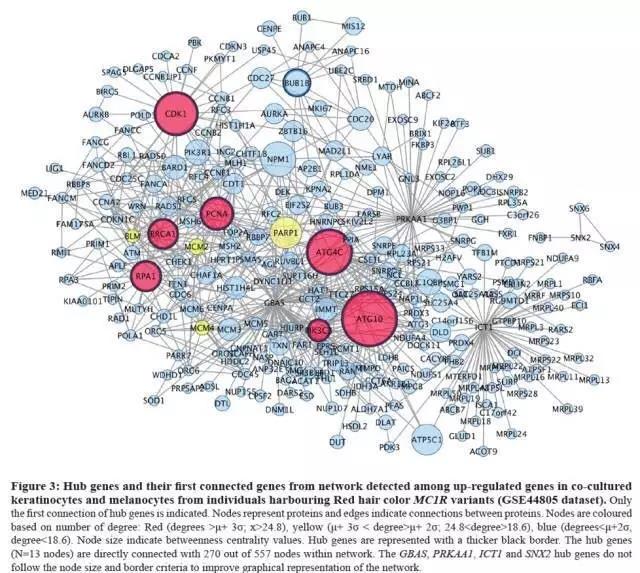
Abstract 里说到 MC1R 这个基因的突变会导致高患癌率的 RHC 表型两种不同的表型,其中 RHC 表型会增加皮肤癌的发生率,那么 MC1R 的突变究竟影响了哪些基因?文章通过 PPI 网络分析,分别对比分析两个不同表型(RHC 和 BHC)的正常皮肤细胞和癌细胞中的差异基因。结果表明,在癌细胞的对比中没有差异,而在正常皮肤细胞中筛选出 23 个 hub 基因,并且其中 8 个基因异常表达,这一结果提示这 8 个基因的异常表达可能是 RHC 表型患癌风险提高的重要原因。
这篇文章利用了 3 个数据包进行综合分析,从而得到了一个 novel 的结论,文章利用 GSE44805 中的差异基因构建 PPI 网络筛选 hub 基因,再利用别的数据包中的测序结果验证这些基因确实存在异常表达,多方验证说明自己生信分析结果是可靠的。虽然作者一点实验也没有做,但是从数据量还有可靠性上来说,可能比自己辛辛苦苦地做小样本量测序还要靠谱。
文章中的分析方法(差异基因以及 PPI 分析)都是我们非常熟悉的。筛选出差异基因,将上调和下调的基因分别构建 PPI 网络,得到文中的 4 张图(不管怎么说,这图的颜值比上一期套路中分析的文章要高得多)。
这张图的构建方法这里不再赘述。
这篇文章的方法完全是可以借鉴和复制的,难点在于找到足够多的具有相似性和可比性的数据结果,以及找到一个合适的切入点得到一个相对 novel 的结论。
下面看 Journal of Proteome Research 上的这篇文章「Weighted Protein Interaction Network Analysis of Frontotemporal Dementia」。
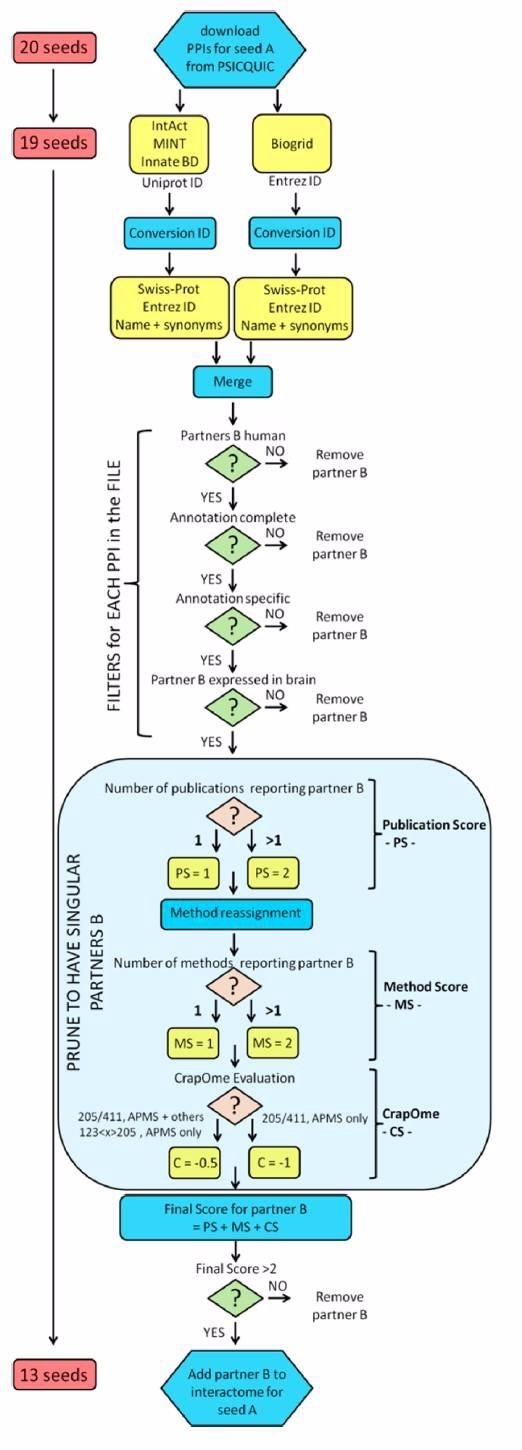
一看这流程图就觉得这文章是生信专业的人做的文章。(本宫上学的时候,就觉得我们生命学院的学生都是码农,生物信息专业、生物医疗工程、生物科学这些专业的人天天都在编代码,完全感受不出生物专业的气息。)
这文章讲得啥咧,就是先选出 13 个种子基因,然后根据 PPI 数据库中蛋白质互作关系构建这 13 个种子基因的第一层网络结构。
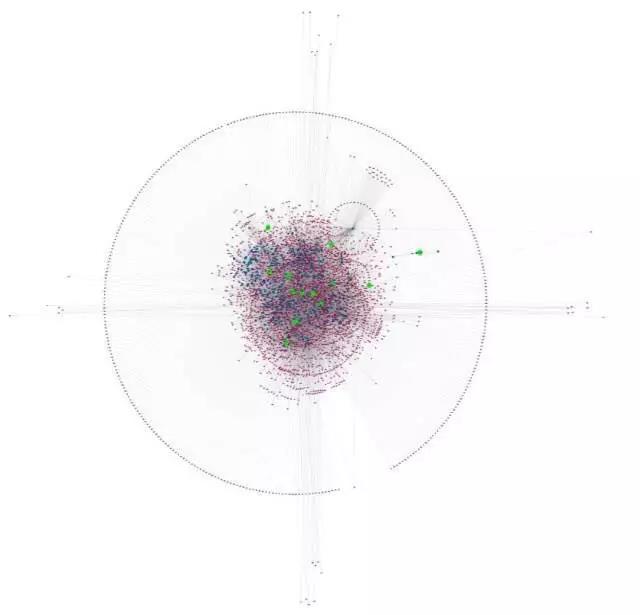
再以第一层网络为种子构建第二层网络结构(然后电脑就死机了)。
然后分析第二层网络的拓扑学结构,从中筛选出 hub 基因(图中绿点表示最初的 13 个种子基因,蓝点表示第一层的基因)。在构建过程中,随着基因数量的不断增加,最先选出的 13 个种子基因未必就是后来的 hub 基因。文中还设置了对照组,并详细讲述了这 13 个种子基因的筛选方法。因为整个分析过程都是建立在生信分析的基础上,属于完全架空的,所以整个研究过程十分讲究逻辑上的严谨性。
之所向大家介绍这篇文章,是觉得这种思路在生信分析的文章中可以借鉴,种子基因的选择可以通过临床上疾病中基因突变的概率来进行筛选,然后构建两层 PPI 网络,进行 GO,KEGG 分析,从而预测新的未知的疾病相关基因,如果后续能从别的数据包中得到表达量的验证或者是自己在临床样本中进行验证,那么整个文章的内容将会更加丰富。
局限性:PPI 数据库中其实很多蛋白质互作结果是没有意义的,因为在实际生物体中很多蛋白质互作情况是不可能发生的,只有在实验人为干预情况下才会发生。